Percentage Correct classification : 95.348837%
Percentage Incorrect classification : 4.651163%
2.9.6. Crab classification
Данный пример демонстрирует использование нейронных сетей для определения пола крабов исходя из их физического размера. Рассматриваются шесть физических характеристик краба: вид, перед, задняя ширина, длина, ширина и размер. Задача – определить пол краба, учитывая наблюдаемые значения каждой из этих шести характеристик.
fid = fopen('private/crabdata.csv');
C = textscan(fid,'%f%f%f%f%f%f%s','delimiter',','); % Import data
fclose(fid);
physchars = [C{1} C{2} C{3} C{4} C{5} C{6}]; % inputs to neural network
female = strncmpi(C{7}, 'Female', 1);
male = strncmpi(C{7}, 'Male', 1);
sex = double([female male]); % targets for neural network
physchars = physchars';
sex = sex';
rand('seed', 491218382)
net = newff(physchars,sex,20); % Create a new feed forward network
[net,tr] = train(net,physchars,sex);
testInputs = physchars(:,tr.testInd);
testTargets = sex(:,tr.testInd);
out = sim(net,testInputs); % Get response from trained network
[y_out,I_out] = max(out);
[y_t,I_t] = max(testTargets);
diff = [I_t - 2*I_out];
f_f = length(find(diff==-1)); % Female crabs classified as Female
f_m = length(find(diff==0)); % Female crabs classified as Male
m_m = length(find(diff==-2)); % Male crabs classified as Male
m_f = length(find(diff==-3)); % Male crabs classified as Female
N = size(testInputs,2); % Number of testing samples
fprintf('Total testing samples: %d\n', N);
cm = [f_f f_m; m_f m_m] % classification matrix
Total testing samples: 40
cm =
18 0
1 21
cm_p = (cm ./ N) .* 100 % classification matrix in percentages
fprintf('Percentage Correct classification : %f%%\n', 100*(cm(1,1)+cm(2,2))/N);
fprintf('Percentage Incorrect classification : %f%%\n', 100*(cm(1,2)+cm(2,1))/N);
cm_p =
45.0000 0
2.5000 52.5000
Percentage Correct classification : 97.500000%
Percentage Incorrect classification : 2.500000%
Во время проведения данной демонстрации не пострадало ни одного краба.
2.9.7. Gene expression analysis
Этот пример демонстрирует поиск образцов в генетических профилях пекарских дрожжей при помощи нейронных сетей.
Целью является получение некоторого представления о Saccharomyces cerevisiae, также известных, как пекарские дрожжи или пивные дрожжи. Это хлеб, который использовался для выпечки хлеба и брожения вина.
Данный пример использует данные микромассива ДНК для изучения временных генетических выражений в практически всех генах Saccharomyces cerevisiae во время «диоксидной смены».
load yeastdata.mat
numel(genes)
ans =
6400
genes{15}
ans =
YAL054C
emptySpots = strcmp('EMPTY',genes);
yeastvalues(emptySpots,:) = [];
genes(emptySpots) = [];
numel(genes)
ans =
6314
nanIndices = any(isnan(yeastvalues),2);
yeastvalues(nanIndices,:) = [];
genes(nanIndices) = [];
numel(genes)
ans =
6276
mask = genevarfilter(yeastvalues);
% Use the mask as an index into the values to remove the filtered genes.
yeastvalues = yeastvalues(mask,:);
genes = genes(mask);
numel(genes)
ans =
5648
[mask, yeastvalues, genes] = genelowvalfilter(yeastvalues,genes,...
'absval',log2(3));
numel(genes)
ans =
822
[mask, yeastvalues, genes] = geneentropyfilter(yeastvalues,genes,...
'prctile',15);
numel(genes)
ans =
614
yeastvalues = mapstd(yeastvalues'); % Normalize data
pc = processpca(yeastvalues,0.15); % PCA
figure
scatter(pc(1,:),pc(2,:));
xlabel('First Principal Component');
ylabel('Second Principal Component');
title('Principal Component Scatter Plot');
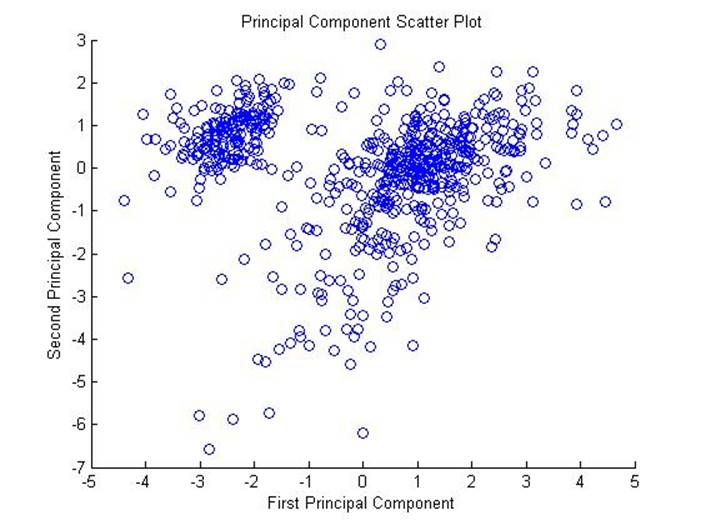
анализ кластеров:самоорганизующиеся карты:
net = newsom(pc,[5 3]);
net = train(net, pc);
figure
plot(pc(1,:),pc(2,:),'.g','markersize',20)
hold on
plotsom(net.iw{1,1},net.layers{1}.distances)
hold off
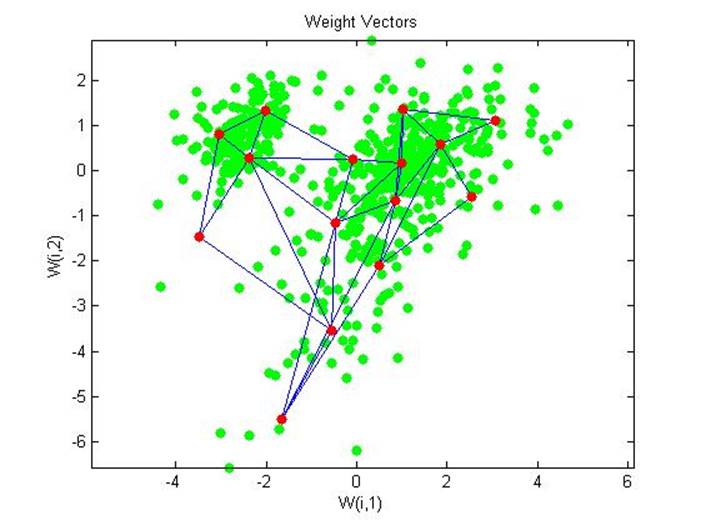
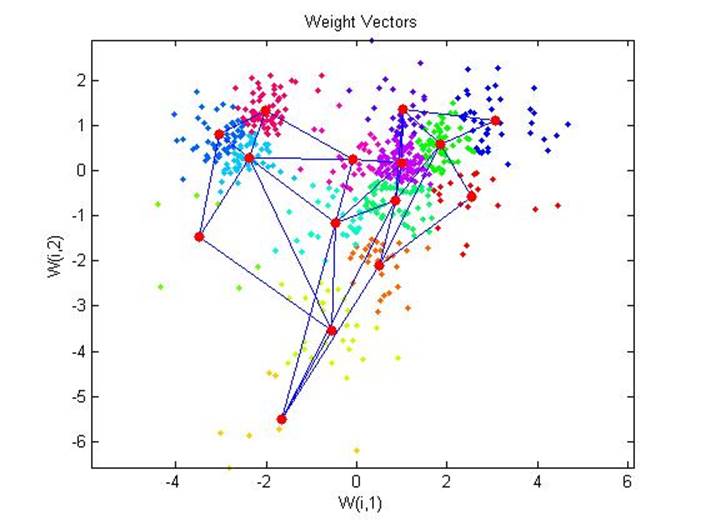
distances = dist(pc',net.IW{1}');
[d,cndx] = min(distances,[],2);
% cndx gives the cluster index
figure
gscatter(pc(1,:),pc(2,:),cndx); legend off;
hold on
plotsom(net.iw{1,1},net.layers{1}.distances);
hold off
2.10. Системы управления
2.10.1. Predictive control of a tank reactor
Уважаемый посетитель!
Чтобы распечатать файл, скачайте его (в формате Word).
Ссылка на скачивание - внизу страницы.